Миграция является фундаментальным свойством клеток, участвующих в поддержании гомеостаза, регенерации, ангиогенезе и иммунной защите организма. В качестве маркера, позволяющего обнаруживать клетки донора в организме реципиента, часто используют т.н. репортерные гены (последовательности, позволяющие изучать локализацию конкретных молекул внутри биологических объектов, например sry-ген Y-хромосомы. Миграционная активность и характер распределения клеток после трансплантации зависит от множества факторов, таких как тканевая принадлежность трансплантированных клеток, количество введенных клеток, путь введения клеток, тип трансплантированных клеток и их микроокружение [1, 2, 3, 4, 7]. Изучение миграции и распределения клеток после трансплантации является обязательным этапом при разработке клеточной терапии различных заболеваний Правильный выбор клеточного материала является определяющим критерием эффективности клеточной терапии. Методы исследования миграции клеток в последнее время интенсивно развиваются и совершенствуются [1, 2, 3]. Для выявления закономерностей миграционной активности и распределения трансплантированных клеток различной степени дифференцировки, полученных на разных временных интервалах после трансплантации и в разных условиях эксперимента, возникает необходимость одновременного анализа как можно большего количества образцов общей ДНК, выделенных из различных органов животных в соответствующие периоды времени. В случае PCR, результаты которого оцениваются полуколичественно по электрофореграмме в денситометре Geldok (Bio-Rad) программой Quantity One или RT-PCR методом на Authorized Termal Cycler – Light Cycler 480 II/96 (Roche), число образцов для одновременного анализа ограничено. Одновременность анализа очень большого числа образцов, в идеале охватывающего весь эксперимент по миграции, позволит избежать ошибок, вносимых при многократном повторении анализа.
Таким образом, разработка множественного, одномоментного и универсального метода анализа распределения клеток является актуальной задачей.
Цель работы заключалась в создании микрочиповой методики количественного анализа распределения клеток костного мозга донора в организме реципиента, оценки путей их миграции, а также в сравнительном анализе данных, полученных микрочиповым методом, с данными электрофореграммы после двухраундовой ПЦР-амплификации и данными ПЦР в режиме реального времени. Для реализации цели необходимо создание унифицированной методики количественного определения ампликонов генов-мишеней, основанной на технологии микрочипового анализа.
Материалы и методы исследования
В работе использовались мыши-самцы и самки линии С57Вl6 в возрасте 8–12 недель. Животные находились на стандартной сбалансированной диете в виварии СО РАМН. Эксперименты на животных проводили в соответствии с соблюдением принципов гуманности, изложенных в директивах Европейского сообщества (86/609/ЕЕС) и Хельсинкской декларации, в соответствии с «Правилами проведения работ с использованием экспериментальных животных». Суспензию костного мозга выделяли из бедренных костей самцов. В стерильных условиях удаляли эпифизы бедренных костей, а диафизы промывали средой RРМI-1640. Клетки костного мозга мышей самцов суспендировали в среде RРМI-1640 при комнатной температуре. Введение суспензии клеток от самцов-доноров мышам-реципиентам (самкам С57Вl6) осуществлялось в хвостовую вену в концентрации 10х106 клеток/мышь.
У сингенных самок реципиентов после трансплантации клеток костного мозга выделялись паховые лимфатические узлы, кожа, печень, селезенка, костный мозг, сердце. Выделенные органы замораживались при температуре – 70 °С.
Проводили выделение ДНК из органов реципиента и определение ее концентрации. ДНК, содержащую маркер трансплантированных клеток (специфическую последовательность Y-хромосомы), выделяли при помощи стандартной обработки додецилсульфатом натрия (SDS) с протеиназой К и методом высаливания [6].
Чистоту и концентрацию выделенной ДНК определяли при помощи спектрофотометра SmartSpecTMSpectrophotometer (Bio-Rad Laboratories, США). Отношение поглощения при длинах волн 260 нм и 280 нм (260 нм/280 нм) показывает чистоту образца ДНК. Для дальнейших исследований были использованы образцы ДНК с отношением A260/A280 не менее 1,7. Для количественного анализа использовалась одинаковая концентрация всех образцов ДНК. Образцы ДНК до начала исследования были заморожены при – 20 °С.
Проводили изучение миграции и распределения трансплантированных клеток костного мозга в организме сингенного реципиента.
Клетки костного мозга ресуспендировали в среде RРМI-1640 при комнатной температуре и вводили мышам-реципиентам (самкам) в хвостовую вену в концентрации 10х106 клеток/мышь. Через один час, 24 часа, 1 месяц и 3 месяца после трансплантации у реципиентов выделяли лимфатические узлы, селезенку, печень, сердце, кожу, костный мозг и замораживали при температуре –70 °С. Для полуколичественного анализа накопления маркера в исследуемых органах проводили методом полимеразной цепной реакции (ПЦР).
Для обнаружения клеток донорского происхождения в органах реципиента использовали маркер Y-хромосомы (ген srу), который был выявлен с помощью полимеразной цепной реакции (ПЦР). Пробы ДНК приводили к единой концентрации, амплифицировали в реакционной смеси, содержащей мышиные праймеры, специфичные для Y-хромосомы [8]. Условия ПЦР: денатурация 95 °С – 3 мин; затем 35 циклов по 50 с при 94 °С, 50 с при 52 °С и 50 с при 72 °С. Далее проводили ПЦР, используя вложенные праймеры [10]. В качестве позитивного и негативного контроля использовали ткань интактных самцов и самок соответственно. Определение маркера в органах проводилось при помощи программного обеспечения Quantity One в денситометре Geldok (Bio-Rad) в единицах оптической плотности ампликонов электрофореграммы.
Проводили полимеразную цепную реакцию с детекцией по конечной точке и ПЦР в режиме реального времени. Для изучения миграции и распределения трансплантированных клеток был использован генетический маркер – специфическая последовательность У-хромосомы (ген Zfy), который был выявлен с помощью полимеразной цепной реакции (ПЦР). Пробы ДНК приводили к единой концентрации, амплифицировали в реакционной смеси, содержащей мышиные праймеры, специфичные для Y-хромосомы [8]. Условия ПЦР: денатурация 95 °С – 3 мин; затем 35 циклов по 50 с при 94 °С, 50 с при 52 °С и 50 с при 72 °С. Далее проводили ПЦР, используя вложенные праймеры [10]. В качестве позитивного и негативного контроля использовали ткань интактных самцов и самок соответственно. Продукты амплификации разделяли электрофорезом в агарозном геле, фотографировали окрашенную интеркалирующим красителем бромистым этидием электрофореграмму в УФ-свете. Интенсивность свечения, которая прямо пропорциональна концентрации маркера в исследуемых органах, оценивали при помощи программного пакета Quantity One в денситометре Geldok (Bio-Rad). Данная программа осуществляет анализ изображений, конвертируя сигналы от биологических образцов в цифровой формат.
Сигнал каждого ампликона содержит, помимо целевого компонента, сигнал фона. Разделение этих компонентов является важнейшим этапом анализа и проводится с помощью компьютерных программ, математических преобразований. Интенсивность флуоресценции для каждого ампликона рассчитывалась как:
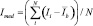 ,
,
где Ii – относительная интенсивность пикселя в пределах области; Ib – средняя относительная интенсивность фоновой флуоресценции. Величины Imed – это усредненные значения флуоресценции ампликона, выражаются в относительных единицах (отн.ед.) оптической плотности.
ПЦР в реальном времени выполнена на Authorized Termal Cycler – Light Cycler 480 II/96 (Roche). Для амплификации целевой последовательности были использованы следующие последовательности праймеров: 5`- TCCAGGCTGGTCGCAAACTCATTT-3`, 5`- ACATGAACAACGCCTTGGGCTTCA -3` и зонд 5`- [FAM]- tccactggcctgtgttggcattgcagttat-<BHQ1>-3`. Реакционная смесь объемом 20 мкл содержала 20 пмоль каждого праймера, 20 пмоль зонда, 5 мкл ДНК. Условия реакции амплификации: денатурация 95 °C в течение 5 минут, 40 циклов: денатурация 95 °C 20 с, отжиг праймеров 65 °C 20 с и элонгация 72 °C 20 с. Для определения абсолютного количества маркерного гена в образцах использовали серию разведений стандарта, в качестве которого была использована плазмида с наработанным геном Zfy Y-хромосомы. Результаты представлены в виде количества трансплантированных клеток на 100 тысяч клеток реципиента. Поскольку в клетках самца содержится по 2 копии гена Zfy Y–хромосомы, количество трансплантированных клеток в образце рассчитывалось по формуле:
N = A/2,
где N – количество Y-позитивных клеток, А – количество гена, определенного в образце. В реакцию брали ДНК образцов одинаковой концентрации, из 100 тыс. клеток.
В качестве внутреннего контроля был использован ген «домашнего хозяйства» β-актина, присутствующий в одинаковых количествах во всех клетках.
Для разработки микрочиповой методики анализа распределения трансплантированных клеток в организме реципиента ампликоны, полученные после двухраундовой ПЦР, очищались от компонентов буфера и праймеров на колонках (Promega, UK). В каждую пробу добавлялся нормировочный олигонуклеотид (NORM, табл. 1). Пробы с общей ДНК наносились на стандартные слайды для микрочипового анализа с аминомодифицированной поверхностью. Печать слайдов осуществлялась контактным методом на споттере SpotArray 24 Microarray Printing System (Perkin Elmer, США). Ковалентную иммобилизацию ДНК-проб на поверхности слайда проводили фотохимически, путем облучения слайдов в течение одной мин в UV-диапазоне (l = 240 нм). Обработка слайдов осуществлялась по методике, описанной в [9]. Для получения флуоресцентной гибридизационной картины микрочипа проводилась гибридизация с использованием олигонуклеотида, модифицированного по 5’- концу красителем Сy3 с добавлением нормировочного олигонуклеотида, модифицированного красителем Cy5 (табл. 1). Гибридизация и отмывка микрочипа проводилась по методике, описанной в [9]. Сканирование микрочипа осуществлялось с использованием конфокального сканера ScanArray Express (Perkin Elmer) на двух длинах волн возбуждения флуоресценции (l = 543 и 633 нм) для получения флуоресцентной картины гибридизации и нормировки флуоресценции соответственно. Количественный анализ интенсивности свечения спотов микрочипа проводился с помощью стандартного программного обеспечения ScanArray Express (Perkin Elmer).
Таблица 1
Последовательности оснований и тип модификации гибридизационного (1), нормировочных (2, 3) и контрольных (4, 5) олигонуклеотидных зондов, использованных в работе
|
№ п/п |
Наименование олигонуклеотида |
Последовательность 5’-3’ |
|
1 |
M_Rev_Cy3 |
Cy3-(CH2)6-5’-GACTCCTCTGACTTCACTTG-3’ |
|
2 |
NORM |
H2N-(CH2)6-5’-TTGGCAGAAGCTATGAAACGATATGGG-3’ |
|
3 |
NORM_Cy5 |
Cy5-(CH2)6-5’-CCCATATCGTTTCATAGCTTCTGCC-3’ |
|
4 |
Q4 |
H2N-(CH2)6 – 5’-ATATCAGATCGCAGTGTA-3’ |
|
5 |
Q4_Cy5 |
Cy5-(CH2)6-5’-TACACTGCGATCTGATAT-3’ |
Примечание. Сy3, Cy5 – цианиновые флуоресцентные метки.

А Б
В
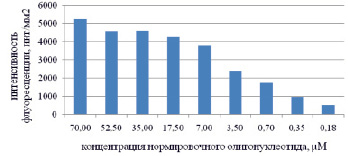
Рис. 1. А) Дизайн чипа – распределение различных концентраций нормировочного олигонуклеотида Q4 на микрочипе; Б) Флуоресцентная картина, полученная в результате гибридизации и сканирования микрочипа с напечатанной калибровочной линейкой; В) Зависимость интенсивности флюоресценции от концентрации нормировочного нуклеотида Q4
Статистическая обработка полученных результатов.
Статистическая обработка результатов проводилась при помощи программы Statistica 6,0 (StatSoft, США). Полученные данные проверяли на нормальность распределения согласно критериям Колмогорова-Смирнова. Наличие и достоверность различий между выборочными величинами независимых выборок оценивали при помощи непараметрического критерия Крускала-Уоллиса. Различия считали достоверными при p < 0,01. Для оценки достоверности различий использовался непараметрический критерий Манна-Уитни. Результаты выражались в единицах оптической плотности (nt/mm) ± стандартное отклонение (M ± σ).
Результаты исследования и их обсуждение
На начальных этапах разработки биочипа для оценки миграционной активности изучаемых клеток проводили количественную оценку насыщаемости поверхностных аминогрупп слайда при иммобилизации контрольного олигонуклеотида (Q4).
Оценка насыщаемости поверхностно-активных функциональных групп была выполнена путем печати контрольного слайда, состоящего из строк с различной концентрацией Q4 (рис. 1, А). Затем проводилась гибридизация с олигонуклеотидом Q5 (100 mM), флуоресцентная картина которой представлена на рис. 1, Б, а количественный анализ приведен в виде гистограммы на рис. 1, В. В результате этого эксперимента был определен диапазон рабочей концентрации контрольных олигонуклеотидов (0,35 µM < CQ4 < 25 µM), пригодный для количественного анализа.
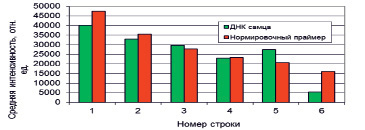
Количественный анализ осуществляется методом «Adaptive circle», входящим в программное обеспечение сканера. Модельный микрочиповый анализ специфической последовательности Y-хромосомы (ДНК самца) разной концентрации, иммобилизованной на аминослайды, проводили с нормировкой по внешнему стандарту. Дизайн микрочипа для отработки методики полуколичественного определения ДНК самца в различных концентрациях показан на рис. 2. В качестве «внешнего» стандарта был взят нормировочный олигонуклеотид NORM (табл. 1). Массив спотов с различной концентрацией ДНК-проб содержал также одинаковое количество олигопробы NORM для нормировки флуоресценции с целью возможности выполнения количественного анализа. На изображении, полученном по каналу Cy5 (рис. 3, А), видна флуоресценция всех спотов микрочипа с обеих сторон, что обусловлено гибридизацией олигонуклеотидов NORM и NORM_Cy5 на сторонах А, В и олигонуклеотида М_Rev_Cy3 и ДНК-пробы на стороне А. Расчет интенсивности флуоресценции стороны А осуществлялся по встроенному алгоритму «Adaptive circle». Результаты количественного анализа приведены на рис. 3, Г.
Из рис. 3, Г, видно удовлетворительное количественное соответствие интенсивности свечения спотов с матричной контрольной ДНК и спотов «внешнего стандарта». Уменьшение концентраций ДНК в исходной смеси и изменение концентрации нормировочного олигонуклеотида привело к скоррелированному уменьшению интенсивностей флуоресценции ДНК-проб и нормировочного олигонуклеотида.

Рис. 2. Дизайн чипа для полуколичественного определения ДНК самца в различных концентрациях методом микрочипового анализа



А Б В
Г
Рис. 3. А, Б, В) флуоресцентная картина, полученная в результате гибридизации и сканирования микрочипа с напечатанной калибровочной линейкой, Б) – канал-Cy3, В) – канал-Cy5, Г) – совмещенное изображение; Д) Зависимость интенсивности флуоресценции от концентрации олигонуклеотида NORM и образцов ДНК самца
Таким образом, выполнена количественная калибровка нормировочных олигонуклеотидов и показано, что рабочий диапазон концентрации нормировочного олигонуклеотида в смеси, наносимой на микрочип, составляет 0,3–25 μМ, при дальнейшем увеличении концентрации происходит насыщение поверхности аминослайда. Разработанная методика подходит для множественного и одномоментного полуколичественного скрининга миграционной активности трансплантированных клеток.
Таблица 2
Наименования ПРЦ-проб в соответствующие периоды времени инкубации самок мыши
|
№ п/п |
Название пробы |
Период отбора |
|
1 |
1ho1 |
Через 1 час, опыт первый |
|
2 |
1so1 |
Через 1 сутки, опыт первый |
|
3 |
1mo1 |
Через 1 месяц, опыт первый |
|
4 |
3mo1 |
Через 3 месяца, опыт первый |
|
5 |
1ho2 |
Через 1 час, опыт второй |
|
6 |
1so2 |
Через 1 сутки, опыт второй |
|
7 |
1mo2 |
Через 1 месяц, опыт второй |
Проводили определение временной картины миграции клеток костного мозга самца мыши в тимусе самки с помощью микрочипового анализа. Для анализа использовалась общая ДНК мыши, выделенная из тимуса самки. Выделение ДНК осуществлялось через час, 24 часа и через месяц после инъекции клеток костного мозга (ККМ) самца (табл. 2), согласно методике, описанной выше. В каждом случае осуществлялась двухраундовая ПЦР с нормированным количеством исходной матрицы – общей ДНК. ПЦР осуществлялась одновременно для всех проб, после окончания всего периода инкубации. Пробы, полученные после второго раунда ПЦР-амплификации, использовались для установления содержания целевого ампликона методом микрочипового анализа.
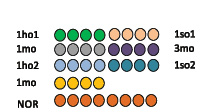
Дизайн микрочипа, используемого для анализа, показан на рис. 4, а. В качестве контроля при микрочиповом анализе использовался олигонуклеотид NORM (табл. 1), который добавлялся в одинаковых количествах во все пробы, подготовленные для печати. Строка 5 чипа содержала только нормировочный олигонуклеотид для количественного определения ампликонов с использованием калибровочной методики, описанной выше.
Дизайн микрочипа позволяет получить одновременную картину распределения ККМ самца в тимусе самки мыши на всех этапах мониторинга (табл. 2) с использованием SRY-гена самца в качестве маркера.
Флуоресцентная картина гибридизации приведена на рис. 4, б (гибридизация M_Rev_Cy3 с ДНК-пробой, канал Cy3), в (гибридизация нормировочных олигонуклеотидов, канал Cy5).
Из рисунка видно достаточно интенсивное свечение всех спотов, полученных по каналу-Cy3 и по каналу-Cy5 с нормировочным олигонуклеотидом. Для сепарации флуоресценции гибридицации целевого ампликона с олигопробой M_Rev_Cy3 от флуоресценции, вызванными возможными неспецифическими взаимодействиями, был предложен следующий метод количественного анализа. Общие интенсивности свечения спотов были рассчитаны по алгоритму [5] с использованием канала-Cy5 в качестве контроля. Далее осуществлялся расчет среднеквадратичного значения флуоресценции для линии из четырех повторов для каждой пробы (рис. 4, а) по формуле:
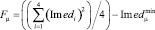 , (4)
, (4)
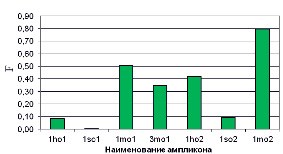
где μ – линия из 4-х повторов, относящихся к конкретному ампликону из соответствующей ДНК пробы (табл. 2), 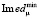 – минимальное значение среднеквадратичной флуоресценции из всех анализируемых линий чипа. Гистограмма, построенная по формуле (4), показана на рис. 5.
– минимальное значение среднеквадратичной флуоресценции из всех анализируемых линий чипа. Гистограмма, построенная по формуле (4), показана на рис. 5.
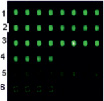
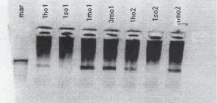
Из рисунка видно удовлетворительное соответствие результатов мониторинга, полученного микрочиповым и электрофоретическим методами. Положение маркера на электрофореграмме соответствует целевому ампликону, а интенствности свечения ампликонов удовлетвортельно коррелируют с данными гисограммы F (рис. 5, а)
Таким образом, впервые выполнена пилотная разработка микрочиповой методики для анализа миграционной активности трансплантированных клеток, позволяющая осуществить одномоментный анализ всех проб, взятых в ходе эксперимента. Методика позволяет расширить дизайн микрочипа для одновременного анализа очень большого числа проб (до нескольких тысяч), что невозможно ни в случае простой ПЦР с последующим электрофоретическим методом анализа, ни в случае ПЦР в режиме реального времени.
Проводили сравнительную оценку результатов исследования миграции и распределения клеток с использованием микрочипового анализа, анализа флуоресценции электрофореграммы и данных, полученных при помощи ПЦР в режиме реального времени. В качестве маркера миграции трансплантированных клеток использована специфическая последовательность Y-хромосомы. Для этого клетки костного мозга самцов внутривенно вводили самкам-реципиентам. Были сравнены результаты количественного анализа распределения клеток в организме-реципиента через один час после трансплантации клеток костного мозга. Анализировались следующие органы: печень, селезенка, костный мозг, лимфоузлы, кожа, сердце.
Для анализа результатов электрофоретическим и микрочиповым методом, осуществлялась двухраундовая ПЦР с нормированным количеством исходной матрицы (ДНК органов самки-реципиента). Визуализация и полуколичественный анализ ампликонов, полученных после второго раунда ПЦР-амплификации, проведены методом микрочипового анализа и электрофоретическим методом. Для ПЦР в режиме реального времени использовались те же образцы ДНК. На рис. 6 показаны результаты всех трех методов.

а) б) в)
Рис. 4. Дизайн микрочипа (а) и картины гибридизации M_Rev_Cy3 с ДНК-пробой (б) и пары нормировочных олигонуклеотидов (в)
а)
б)
Рис. 5. Результаты микрочипового анализа распределения ККМ самца мыши в пробах, выделенных из тимуса самки на различных этапах мониторинга (табл. 2) (а), и соответствующая электрофоретическая картина (б)
А
Б
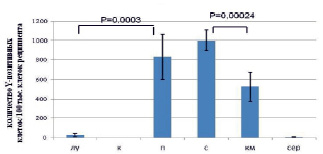
В
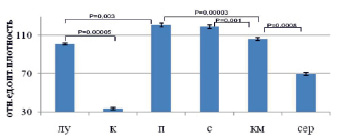
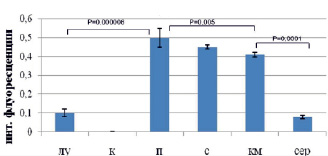
Рис. 6. Результаты анализа количества Y-позитивных клеток в органах реципиента после сингенной трансплантации клеток самца самкам-реципиента методом анализа результатов ПЦР электрофоретическим способом (А), микрочиповым способом (Б), ПЦР в режиме реального времени (В). Примечания: лу – лимфатические узлы, к – кожа, п – печень, с – селезенка, км – костный мозг, сер – сердце
Показано, что максимальное количество Y-позитивных клеток определено в печени, селезенке и костном мозге тремя способами, минимальное – в коже реципиента (рис. 6). Всеми тремя способами показано, что в лимфатических узлах маркера трансплантированных клеток больше, по сравнению с сердцем реципиента. Все три метода подходят для описания распределения трансплантированных клеток. ПЦР в режиме реального времени позволяет определить точное количество трансплантированных клеток в органах реципиента, способен детектировать небольшие различия в количестве мигрировавших клеток.
Заключение
Таким образом, показано удовлетворительное качественное соответствие результатов микрочипового анализа, результатов анализа электрофореграммы и данных, полученных при помощи ПЦР в режиме реального времени. Данные, полученные при помощи микрочиповой методики, совпадают с результатами опыта, полученными с использованием других методов. Преимуществом предложенного биочипового метода является одномоментность и множественность анализа, что позволяет проанализировать несравнимо большее количество проб, уменьшить уровень экспериментальных ошибок из-за многократного повторения анализа.



