Качество распознавания отдельных объектов изображения, значительно зависит от этапа сегментации. Существующие методы предусматривают полуавтоматический режим определения порогов сегментации с последующим сохранением выбранных значений от препарата к препарату. Как правило, эффективное значение порога от препаратов к препарату не сохраняется, что приводит к искажению результатов. Поэтому использование методов со статическими значениями порогов сегментации при автоматизированном анализе нежелательно.
Алгоритмы сегментации цветных изображений позволяют выявлять объекты, отличающиеся по яркостным, цветовым и текстурным характеристикам. Количество настроечных параметров алгоритмов растёт пропорционально количеству разделяемых признаков. Увеличение количества настроечных параметров приводит к потере автоматической составляющей алгоритма, либо требует дополнительных методов для их автоматической коррекции.
Анализ изображений медико-биологических препаратов позволяет сделать вывод о слабо выраженных текстурных признаках объектов изображения, следовательно, для сегментации достаточно оперировать цвето-яркостными характеристиками.
Для сегментации цифровых изображений был выбран алгоритм кластерного анализа с самообучением. Задача кластерного анализа обеспечить редукцию некоторого множества данных в более компактную классификацию объектов.
Для обеспечения качества кластеризации и избежания возможного нахождения псевдо - центров, предложен метод предварительного анализа яркостных зон изображения с целью выявления начального значения центроидов, каждого кластера.
Для оценки качества сегментации использовалась среднеквадратическая ошибка отклонения исходного множества значений от центров кластеров и оценка эксперта (выраженная в процентах отражающих относительное качество детализации).
В качестве количественной метрики кластерной принадлежности было выбрано Евклидово расстояние:
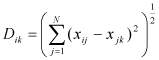
где: i - индекс текущего наблюдения, k - индекс кластера, N - количество признаков цветового пространства (N=3), x={x1,x2,x3} - вектор в выбранном цветовом пространстве.
Более качественные результаты удалось получить после перевода метрик цветового пространства HSV, в декартову систему координат XhsYhsZv .
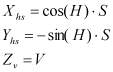
Результаты кластеризации в разных цветовых пространствах представлены в таблице 1. Следует отметить, что исследования проводились также в пространствах YES, PHS, CMYK но из-за неудовлетворительных результатов показатели не внесены в таблицу.
Как видно из таблицы максимально приемлемым можно считать результат кластеризации в пространстве XhsYhsZv.
Таблица 1. Результаты кластеризации для тестового изображения
|
Цветовое пространство |
Среднеквадратическая ошибка отклонения |
Качество сегментации оцененное экспертом |
|
HSL |
19.03 |
70% |
|
HSV |
8.09 |
70% |
|
RGB |
13.25 |
70% |
|
LAB |
0.36 |
75% |
|
XhsYhsZl |
12.7 |
75% |
|
XhsYhsZv |
0.041 |
99% |
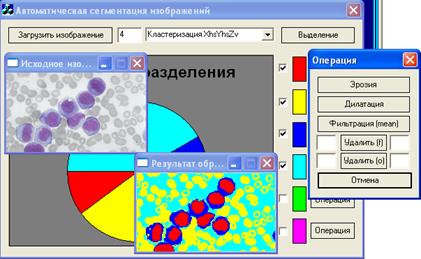
Рис. 1. Программное обеспечение для сегментации изображений медико-биологических препаратов (внешний вид).
В результате работы определенно цветовое пространство обеспечивающее достаточное качество сегментации медико-биологических препаратов методом кластерного анализа. Предложена методика предварительного вычисления значений центроидов. Приведены формулы перевода метрик цветового пространства HSV. Эффективность применения предлагаемого метода заключается в автоматическом выявлении объектов интереса на совокупном изображении, настроечными параметрами алгоритма является число, задающее количество выделяемых сегментов. Для исследования методов на практике было разработано программное обеспечение (см. рис.1).
СПИСОК ЛИТЕРАТУРЫ:
- Грузман И.С., Киричук В.С., и др. Цифровая обработка изображений в информационных системах Учебное пособие. Новосибирск 2000
- Павлидис Т. Алгоритмы машинной графики и обработка изображений. - М.: Радио и связь, 1988.
- Курс лекций по дисциплине «Введение в компьютерную графику". ВМиК МГУ, 2003



